Biophysik
Einfügen und Falten von Membranproteinen
Biologische Membranen sind wesentliche strukturierende Elemente aller lebenden Zellen. Sie bestehen aus einer Phospholipid-Doppelschicht, die eine wasserabweisende Barriere gegen den willkürlichen Austausch von gelösten Stoffen bildet, und membranüberspannenden sowie peripher assoziierten Proteinen. Membranproteine ermöglichen den geregelten Austausch von gelösten Stoffen oder die Weiterleitung von Signalen von einer Seite der Membran zur anderen oder führen enzymatische oder andere Funktionen aus. Obwohl Membranen für alle Lebewesen essenziell sind, ist ihre Biogenese nicht gut verstanden und daher ist dies das Forschungsgebiet für viele Zellbiologen, Biochemiker und Biophysiker. Es gibt nur zwei Typen von Transmembranproteinen, die sich durch die Sekundärstruktur ihrer Transmembrandomänen unterscheiden, nämlich α-helical und β-barrel integrale Membranproteine. Die biophysikalischen Mechanismen ihres Einbaus und ihrer Faltung in Membranen sind nur unzureichend verstanden. Wir haben wesentliche biophysikalische Prinzipien der Faltung und des Membraneinbaus von β-Fass-Membranproteinen aus äußeren Membranen (OM) von Bakterien wie Escherichia coli und auch der OM von Mitochondrien aus Zellen von Homo sapiens beschrieben [1]. Dazu gehören strukturelle Charakterisierungen von Faltungsintermediaten (Abb. 1B) von hVDAC1 und der Transmembrandomäne des Außenmembranproteins A (OmpA, die Aminosäuresequenz von OmpA ist in Abb. 1A gezeigt) und Interaktionen von bakteriellen OMPs (OmpA, OmpG, BamA, FomA, NalP etc.) mit molekularen Chaperonen aus dem bakteriellen Periplasma, wie Skp, SurA und FkpA. Für diese Untersuchungen haben wir eine Reihe von neuartigen Methoden und Ansätzen entwickelt, siehe z. B. [2, 3]
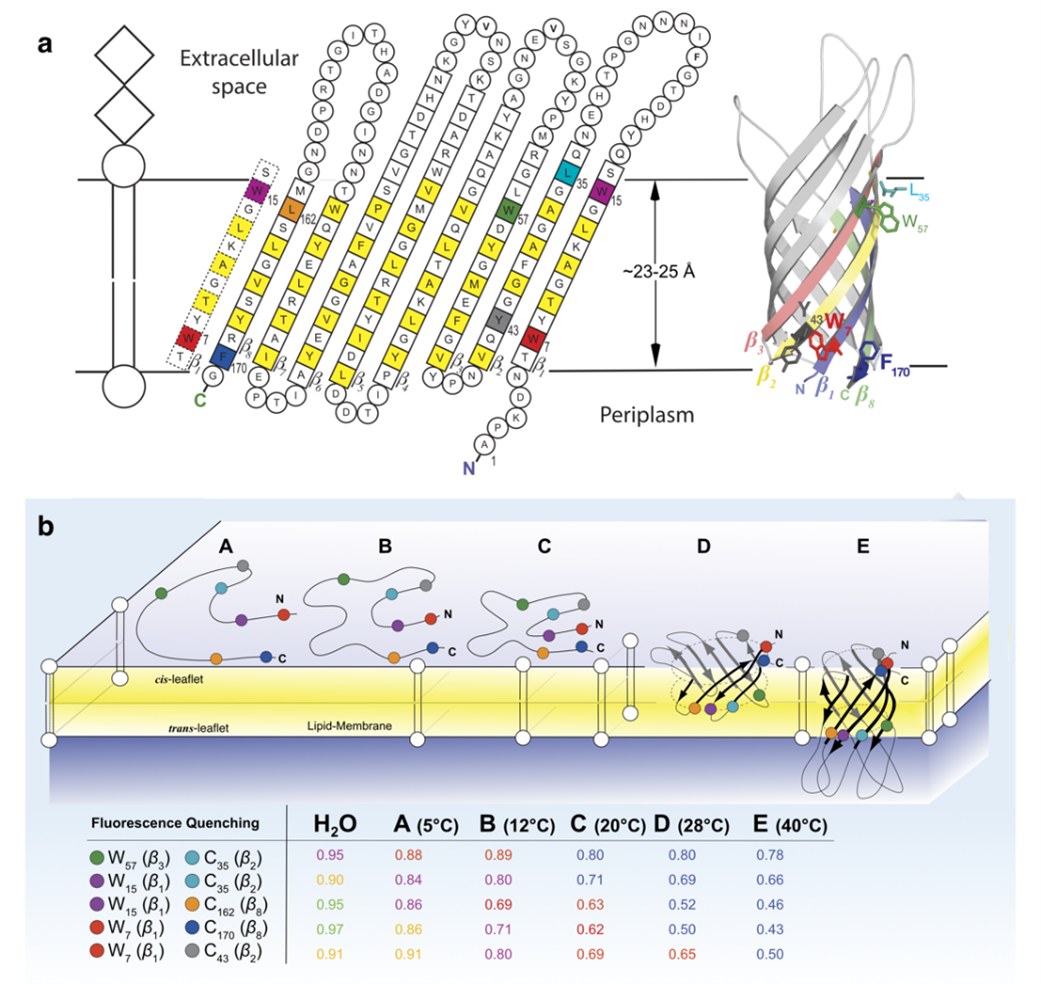
Während die Faltung und der Membraneinbau von β-Fassproteinen in reine Lipiddoppelschichten, Detergens-Mizellen oder auch amphipathische Polymere spontan erfolgen kann, z. B. aus einer denaturierten Form des β-Fass-Membranproteins in Lösungen chaotroper Reagenzien wie Harnstoff, wurde in den letzten Jahren ein heteropentamere Proteinkomplex aus dem OM von E. coli identifiziert, der als Barrel Assembly Machinery (BAM)-Komplex bezeichnet wird und der für den Einbau und die Faltung in zelluläre OMs erforderlich ist. Das OM von E. coli hat eine Lipidzusammensetzung, die eine spontane Faltung und Insertion verhindert. Daher ist der BAM-Komplex essenziell für die Lebensfähigkeit der Zelle. Die physikalischen Prinzipien, nach denen der BAM-Komplex arbeitet, sind derzeit ein Forschungsschwerpunkt der Arbeitsgruppe Biophysik. Wir haben alle fünf Proteine des BAM-Komplexes von E. coli isoliert und ihre Funktion bei der Faltung von Membranproteinen an unserem Hauptmodellprotein für Faltungsstudien, OmpA, untersucht. Nur zwei Untereinheiten des BAM-Komplexes sind als essenziell für das Überleben von E. coli unter Labor Bedingungen bekannt, BamA (~800 Aminosäurereste) und BamD (~240 Reste). Allerdings toleriert E. coli auch die gleichzeitige Eliminierung von BamB und BamE nicht. Auf der anderen Seite enthalten bestimmte Bakterien wie Thermus thermophiles nur BamA, das evolutionär konserviert ist, aber nicht die anderen 4 Untereinheiten, die in E. coli gefunden werden. Diese Beobachtungen deuten darauf hin, dass BamA zentral für die Funktion des BAM-Komplexes aus E. coli ist und dass die anderen 4 Untereinheiten spezialisierte Funktionen erfüllen. Durch biophysikalische Analysen konnten wir kürzlich mehrere Schlüsseleigenschaften des BAM-Komplexes aus E. coli nachweisen. Zu diesen gehören:
- Die drei Hauptkomponenten des BAM-Komplexes, BamA, BamB und BamD, sind alle einzeln in der Lage, die Kinetik der Faltung von OmpA in Modellmembranen zu beschleunigen, was darauf hindeutet, dass diese Untereinheiten eher an aufeinanderfolgenden als an parallelen Schritten beteiligt sind, die eine konzertierte Aktion erfordern.
- Wir konnten ein Membranmodellsystem entwickeln, das es uns erlaubt, Faltungsintermediate von OmpA (und wahrscheinlich von anderen OMPs) gebunden an BamA einzufangen. Dies ermöglicht die Identifizierung ihrer lokalen Strukturen und auch ihrer lokalen Kontakte mit BamA.
- Wir haben direkte Interaktionen des molekularen Chaperons Skp aus dem Periplasma von E. coli mit BamD und BamA identifiziert und charakterisiert.
- Wir haben die Bindungsregionen von BamB an Membranlipide bestimmt und gezeigt, dass BamB nicht nur über seinen N-Terminus an der Membran verankert ist, sondern auch über eine separate Lipidbindungsdomäne.
Dies sind Schlüsselergebnisse, um die Rollen und die Funktionen des gesamten BAM-Komplexes von E. coli sowie seiner einzelnen Untereinheiten zu bestimmen. Sie sind auch eine solide Basis, um BAM-Interaktionen mit verschiedenen OMPs zu untersuchen, die in gramnegativen Bakterien wie E. coli von Bedeutung sind. Dies ist nicht nur für die Grundlagenforschung von Interesse, sondern auch für biotechnologische Anwendungen angesichts einer wachsenden Zahl von multiresistenten Stämmen pathogener gramnegativer Bakterien. Die Grundlagenforschung könnte eine Basis für neue Klassen von Antibiotika liefern, die den BAM-Komplex oder wichtige Client-OMPs dysfunktional machen und so die Bakterien abtöten.
[1] J.H. Kleinschmidt, Biochim Biophys Acta 1848 (2015) 1927-1943.
[2] A. Schüßler, S. Herwig, J.H. Kleinschmidt, Methods Mol Biol 2003 (2019) 145-162.
[3] L. Gerlach, O. Gholami, N. Schürmann, J.H. Kleinschmidt, Methods Mol Biol 2003 (2019) 465-492.
Prof. Dr. Jörg H. Kleinschmidt
Ordentliches Mitglied

- Telefon
- +49 561 804-4041
- jhk[at]uni-kassel[dot]de
- Website
- Biophysik
- Standort
- Universität Kassel
Fachbereich 10 - Naturwissenschaften & Mathematik
Institut für Biologie
Heinrich-Plett-Straße 40
34132 Kassel
- Raum
- 2249
